- 软件gydF4y2Ba
- 开放获取gydF4y2Ba
- 发表:gydF4y2Ba
野兽:贝叶斯进化分析抽样树gydF4y2Ba
BMC进化生物学gydF4y2Ba体积gydF4y2Ba7gydF4y2Ba,文章号:gydF4y2Ba214gydF4y2Ba(gydF4y2Ba2007gydF4y2Ba)gydF4y2Ba
摘要gydF4y2Ba
背景gydF4y2Ba
分子序列变异的进化分析是一项统计工作。这反映在系统发育推断、多序列比对和分子群体遗传学的概率模型的使用增加。在这里,我们提出BEAST:一种快速、灵活的软件架构,用于进化树相关的分子序列的贝叶斯分析。提供了大量流行的序列演化随机模型,并实现了适用于种内和种间序列数据的基于树的模型。gydF4y2Ba
结果gydF4y2Ba
BEAST版本1.4.6包含81000行Java源代码、779个类和81个包。它提供了DNA和蛋白质序列进化模型,高度参数化的联合分析,宽松的时钟系统发育,非同期序列数据,统计比对和广泛的先验分布选项。BEAST源代码是面向对象的,模块化的设计,可在gydF4y2Bahttp://beast-mcmc.googlecode.com/gydF4y2Ba在GNU LGPL许可下。gydF4y2Ba
结论gydF4y2Ba
BEAST是一个强大而灵活的分子序列变异进化分析包。它也为进化分析的新模型和统计方法的进一步发展提供了资源。gydF4y2Ba
背景gydF4y2Ba
进化和统计学是渗透在现代分子序列变异分析中的两个共同主题。现在人们普遍认为,大多数关于分子序列的问题本质上是统计的,应该在参数估计和假设检验方面加以框架。同样,很明显,要建立模型,准确地描述分子序列的变化,进化的角度是必要的。gydF4y2Ba
BEAST软件包是一项雄心勃勃的尝试,旨在从分子序列数据为进化模型的参数估计和假设检验提供一个一般框架。BEAST是一个贝叶斯统计框架,因此为先验知识与数据提供的信息结合提供了一个角色。贝叶斯马尔可夫链蒙特卡罗(MCMC)已经被热情地接受为最先进的方法进行系统发育重建,主要是由快速和广泛的采用gydF4y2BaMrBayesgydF4y2Ba[gydF4y2Ba1gydF4y2Ba].这种热情可以归因于许多因素。首先,贝叶斯方法允许相对简单地实现极其复杂的进化模型。其次,人们经常错误地认为贝叶斯估计比基于最大似然准则的启发式优化“更快”。gydF4y2Ba
除了系统发育推断外,一些研究人员最近开发了贝叶斯MCMC软件,用于从遗传数据中基于合并估计人口统计学参数[gydF4y2Ba2gydF4y2Ba- - - - - -gydF4y2Ba7gydF4y2Ba].像系统发育分析一样,这些也需要底层模型中的基因树,尽管在这种设置中,序列代表来自同一物种的不同个体,而不是来自不同物种。最近,贝叶斯MCMC也被应用于进化生物信息学的一个核心问题:系统发育和序列比对的共同估计[gydF4y2Ba8gydF4y2Ba,gydF4y2Ba9gydF4y2Ba].结合系统发育和基于聚合的群体遗传学的进展,贝叶斯MCMC已被应用于大多数分子数据中常见的进化问题。gydF4y2Ba
BEAST可以与许多其他具有类似目标的软件包进行比较,例如gydF4y2BaMrBayesgydF4y2Ba[gydF4y2Ba1gydF4y2Ba],目前专注于系统发育推断和gydF4y2Ba蝙蝠翼战斗机gydF4y2Ba[gydF4y2Ba4gydF4y2Ba],主要关注基于聚合的微卫星群体遗传学。和这些软件包一样,BEAST实现的核心算法是Metropolis-Hastings MCMC [gydF4y2Ba10gydF4y2Ba,gydF4y2Ba11gydF4y2Ba].MCMC是一种随机算法,它对选择的目标分布产生基于样本的估计。对于我们的目的,目标分布是给定一组分子序列的一组进化参数的后验分布。BEAST最显著的特点可能是它对校准系统发育和系谱的坚定关注,也就是说,包含时间尺度的根树。这是通过显式地模拟树中每个分支的分子进化速度来实现的。在最简单的层面上,这可以是整个树的均匀速率(即分子钟模型[gydF4y2Ba12gydF4y2Ba]),而这个比率是预先知道或根据校正资料估计的。分子系统发育最近最有前途的进展之一是引入了gydF4y2Ba放松分子钟gydF4y2Ba不假设谱系间速率恒定的模型[gydF4y2Ba13gydF4y2Ba- - - - - -gydF4y2Ba20.gydF4y2Ba].BEAST是第一个允许在这种模型下推断实际系统发育树的软件包[gydF4y2Ba21gydF4y2Ba].gydF4y2Ba
BEAST开发背后的目的是将大量互补的进化模型(替换模型、插入-删除模型、人口统计模型、树形先验、放松时钟模型、节点校准模型)带入一个单一的连贯框架,用于进化推断。这种由许多简单模型组件构建复杂进化模型的基本原理为分子序列分析提供了强大的新可能性。这样做的动机是:(1)避免目前存在于许多进化分析包中的不必要的简化假设;(2)提供新的模型组合和灵活的模型规范系统,以便研究人员可以根据他们的特定问题集调整他们的进化分析。gydF4y2Ba
这个项目的雄心需要团队合作,我们希望通过使BEAST的源代码免费可用,实现的模型的范围,虽然已经很大,将继续增长和多样化。gydF4y2Ba
结果与讨论gydF4y2Ba
BEAST在演化模型的规范方面提供了相当大的灵活性。例如,考虑对编码DNA的多个序列比对的分析。在BEAST分析中,可以允许每个密码子位置具有不同的替代率、不同位点之间的不同数量的速率异质性以及不同分支之间的不同数量的速率异质性,同时与其他密码子位置共享相同的转换到反转的内在比率。事实上,任何或所有参数(包括树本身)都可以在序列数据的分区之间共享或独立。gydF4y2Ba
贝叶斯统计分析的一个不可避免的特征是参数值的先验分布的规范。这种要求既是一种优势,也是一种负担。这是一个优势,因为相关知识,如古生物系统发育的校准,很容易纳入分析。然而,当一个参数不存在明显的先验分布时,研究人员就有责任确保先验选择不会无意中影响感兴趣参数的后验分布。gydF4y2Ba
在BEAST中,所有参数(无论是替换的、人口统计的还是系谱的)都可以给出信息先验(例如,指数型、正态、对数正态或有边界的一致,或这些参数的组合)。例如,树的根的年龄可以给定一个具有预先指定的平均值的指数先验。gydF4y2Ba
进化的模式gydF4y2Ba
在BEAST中,一组对齐的核苷酸或氨基酸序列的进化模型分为五个部分。对于其中的每一种都提供了一系列的可能性,因此可以很容易地构建出大量独特的进化模型。这些组件是:gydF4y2Ba
替换模型-替换模型是一个齐次马尔可夫过程,它定义了沿着树的分支发生不同替换的相对速率。gydF4y2Ba
•站点间的速率模型-站点间的速率模型定义了站点间进化变化的相对速率的分布。gydF4y2Ba
•分支之间的速率模型——分支之间的速率模型定义了分支之间的速率分布,并用于将以时间为单位的树转换为替换的单位。这些模型对于估计发散时间是很重要的。gydF4y2Ba
树-序列的系统发育或系谱关系的模型。gydF4y2Ba
•树先验——树先验提供了节点高度(以时间为单位)和树拓扑的参数化先验分布。gydF4y2Ba
站点间的替代模型和费率模型gydF4y2Ba
对于核苷酸数据,所有嵌套在一般时间可逆(GTR)模型中的模型[gydF4y2Ba22gydF4y2Ba,gydF4y2Ba23gydF4y2Ba] -包括著名的HKY85型号[gydF4y2Ba24gydF4y2Ba-可指定。对于氨基酸序列比对的分析,可以使用以下任何替代模型:Blosum62, CPREV, Dayhoff, JTT, MTREV和WAG。当核苷酸数据表示编码序列(即去除内含子的帧内蛋白质编码序列)时,Goldman和Yang模型[gydF4y2Ba25gydF4y2Ba]可以用来模拟密码子进化。此外,各网站的Γ-distributed费率[gydF4y2Ba26gydF4y2Ba,gydF4y2Ba27gydF4y2Ba]或不变地点的比例,或两者的组合[gydF4y2Ba28gydF4y2Ba,gydF4y2Ba29gydF4y2Ba]可以用来描述站点之间的速率异质性。gydF4y2Ba
分支间速率模型,发散时间估计和时间戳数据gydF4y2Ba
BEAST支持的分支间速率的基本模型是严格分子钟模型[gydF4y2Ba12gydF4y2Ba],通过指定替换率或节点或节点集的日期进行校准。在这种情况下,可以估计特定演化支的分化日期。演化支可以被定义为类群的强制分组,也可以被定义为一组感兴趣的类群的最近的共同祖先。第二种选择不要求所选分类单元相对于树的其余部分具有单一性。此外,当与树的顶端相关的日期差异占整个树的年龄的很大比例时,这些日期可以纳入模型,提供关于进化变化总体速率的信息来源[gydF4y2Ba3.gydF4y2Ba,gydF4y2Ba30.gydF4y2Ba,gydF4y2Ba31gydF4y2Ba].gydF4y2Ba
在BEAST中,发散时间估计也被扩展到包括gydF4y2Ba放松系统发生学gydF4y2Ba模型,其中进化的速度允许在树的不同分支之间变化。特别地,我们支持一类不相关的放松时钟分支速率模型,其中每个分支的速率来自于一个潜在的分布,如指数或对数正态分布[gydF4y2Ba21gydF4y2Ba].gydF4y2Ba
如果序列数据都来自于一个时间点,则必须指定具有强先验的整体进化速率。进化速率的先验所暗示的单位将决定树中节点高度的单位(包括最近共同祖先的年龄)以及人口参数的单位,如种群大小参数和增长率。例如,如果进化速率设置为1.0,则节点高度(和根高度)将以每个站点的突变为单位(即由常见软件包产生的分支长度的单位,如gydF4y2BaMrBayesgydF4y2Ba3.0)。类似地,对于单倍体群体,合并参数将是的估计值gydF4y2BaNgydF4y2BaegydF4y2BaμgydF4y2Ba,在那里gydF4y2BaNgydF4y2BaegydF4y2Ba有效种群大小和gydF4y2BaμgydF4y2Ba是每一代的突变率。然而,例如,如果进化速率以每年每个位点的突变表示,那么树中的分支将以年为单位。此外,人口模型的人口规模参数将等于gydF4y2BaNgydF4y2BaegydF4y2BaτgydF4y2Ba,在那里gydF4y2BaτgydF4y2Ba是以年为单位的代长。最后,如果进化速率以每代每个位点的突变单位表示,则生成的树将以代为单位,人口模型的种群参数将以自然单位表示(即将等于繁殖个体的有效数量)。gydF4y2BaNgydF4y2BaegydF4y2Ba).gydF4y2Ba
树先验gydF4y2Ba
当从同质群体中收集序列数据时,各种合并[gydF4y2Ba32gydF4y2Ba,gydF4y2Ba33gydF4y2Ba人口历史模型可以在BEAST中用于模拟人口规模随时间的变化。目前,可用的简单参数模型包括常量尺寸gydF4y2BaNgydF4y2Ba(gydF4y2BatgydF4y2Ba) =gydF4y2BaNgydF4y2BaegydF4y2Ba(1参数),指数增长gydF4y2BaNgydF4y2Ba(gydF4y2BatgydF4y2Ba) =gydF4y2BaNgydF4y2BaegydF4y2BaegydF4y2Ba-gydF4y2BagtgydF4y2Ba(2个参数)和逻辑增长(3个参数)。gydF4y2Ba
此外,高度参数化的贝叶斯天际线图[gydF4y2Ba34gydF4y2Ba]也是可用的,但该模型只能在数据对人口历史有很强的信息时使用。所有这些人口统计模型都是树中节点年龄的参数先验,其中超参数(例如,人口规模,gydF4y2BaNgydF4y2BaegydF4y2Ba,和增长率,gydF4y2BaggydF4y2Ba)可以进行抽样和估计。在同一人口统计模型下,可以同时分析两个或多个未链接的基因树,也可以进行基于单位点合并的推理。复杂的多位点联合推理可以通过为每个位点分配单独的总体速率和替代过程来实现,从而适应具有异质进化过程的位点。gydF4y2Ba
目前关于树形和分枝率的非聚结先验的选择有限。目前,一个简单的圣诞前出生率的新血统(1参数)可以采用。然而,广义的出生-死亡树先验正在研究中。gydF4y2Ba
除了分支时间的一般模型,如合并先验和Yule先验,树先验还可以包括特定的分布和/或对某些节点高度和拓扑特征的约束。这些额外的先验可能代表其他知识来源,如专家对化石记录的解释。例如,如上所述,树中的每个节点都可以具有表示其日期知识的先验分布。这种基于指定一个节点的日期来校准树的方法已经有很长的历史了[gydF4y2Ba35gydF4y2Ba].最近一篇关于“放松系统发育”的论文包含了更多关于校准先验的信息[gydF4y2Ba21gydF4y2Ba].gydF4y2Ba
Insertion-deletion模型gydF4y2Ba
最后,BEAST还有一个插入-删除过程的模型。这提供了共同估计系统发育和多序列比对的能力。目前只有TKF91插入-删除模型[gydF4y2Ba36gydF4y2Ba]可用。有兴趣的读者可参阅有关此主题的论文,以了解更多详情[gydF4y2Ba8gydF4y2Ba].gydF4y2Ba
多个数据分区以及链接和解链接参数gydF4y2Ba
BEAST提供了同时分析多个数据分区的能力。当在单个多位点联合分析中组合多个基因时,这是有用的(例如[gydF4y2Ba37gydF4y2Ba])或将不同的进化过程分配到序列对齐的不同区域(如密码子位置;如。gydF4y2Ba6gydF4y2Ba])。在涉及多个分区的分析中,替换模型的参数、站点之间的速率模型、分支之间的速率模型、树和先验树都可以“链接”或“不链接”。例如在Lemey对HIV-1 O组的分析中gydF4y2Ba等gydF4y2Ba[gydF4y2Ba37gydF4y2Ba],假设三个基因座(gag, int, pol)共享相同的替代模型参数(GTR),以及共享相同的指数增长人口统计历史。然而,它们被假设在站点之间具有不同的Γ-distributed速率异质性的形状参数,严格分子钟的不同速率参数以及三种树拓扑和发散时间集也被假设为独立且不相关。gydF4y2Ba
模型比较和模型选择gydF4y2Ba
在贝叶斯框架中,模型比较最合理的理论框架是贝叶斯因子(BF)的计算:gydF4y2Ba
在哪里gydF4y2BapgydF4y2Ba(gydF4y2BaDgydF4y2Ba|gydF4y2Ba米gydF4y2Ba)为模型M的边际似然,取模型参数值的平均值gydF4y2BaθgydF4y2Ba:gydF4y2Ba
BF是两个模型的边际可能性之比。一般来说,计算BF涉及一个可逆的跳跃MCMC,其中构造了一个马尔可夫链,对包含两个模型的状态空间进行采样。可逆跳跃MCMC还没有在BEAST中实现。然而,通过处理BEAST分析的输出,有几种方法可以近似一个模型的边际似然(因此两个模型之间的BF)。牛顿和拉夫特首先提出了一个简单的方法[gydF4y2Ba38gydF4y2Ba]通过重要性抽样(后验作为重要性分布)计算BF。根据这个重要性分布,抽样似然的调和平均值是边际似然的估计量:gydF4y2Ba
这个估计器并不总是表现得很好,但有许多修改可以用来稳定它,并且可以使用自举来评估估计的边际可能性的不确定性。一般来说,BF > 20是支持比较好的型号(gydF4y2Ba米gydF4y2Ba1gydF4y2Ba在方程gydF4y2Ba1gydF4y2Ba).gydF4y2Ba
例子gydF4y2Ba
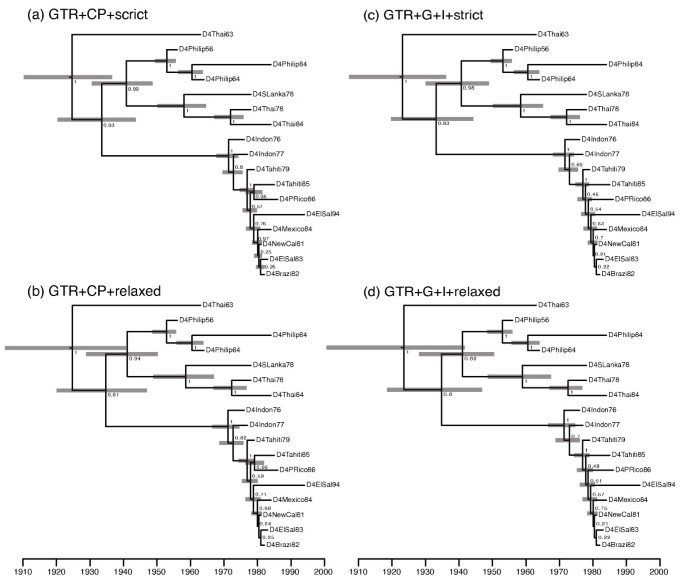
我们展示了对从1956年至1994年分离的17个登革热病毒血清型4序列样本进行贝叶斯分析的一些关键特征(见[gydF4y2Ba30.gydF4y2Ba]查阅详情)。与许多RNA病毒一样,登革病毒的进化速度很快,因此BEAST可以使用17个分离株的采样时间作为校准,以估计总体替代率和多年的分化时间。我们分析了密码子位置特定替代模型(GTR + CP)下的数据,其中每个密码子位置都有一个单独的相对替代率参数,以及标准的GTR + Γ + I模型。这两个模型有相同数量的自由参数。我们还研究了两种不同的分支速率变化模型:严格时钟和不相关的对数正态分布松弛分子钟。我们使用恒定种群规模的合并树作为先验树。对于每个模型,MCMC运行10,000,000步,每500步采样一次。每次运行的前10万步被丢弃为burnin。这导致所有四种分析的后验概率的有效样本量远远大于1000(见附加文件)gydF4y2Ba1gydF4y2Ba,gydF4y2Ba2gydF4y2Ba,gydF4y2Ba3.gydF4y2Ba和gydF4y2Ba4gydF4y2Ba,用于所有四次运行的BEAST XML输入)。gydF4y2Ba
正如先前所指出的,蛋白质编码序列的一般情况[gydF4y2Ba39gydF4y2Ba],我们发现,站点间率异质性的密码子位置特定模型比GTR + Γ + I模型对数据的拟合要优越得多(见表gydF4y2Ba1gydF4y2Ba),并且还支持不同的共识树拓扑(参见图gydF4y2Ba1gydF4y2Ba).然而,我们发现分支之间的两个速率变化模型之间的差异很小(log BF = 0.8),这表明这一特定数据可以像之前建议的那样被视为时钟[gydF4y2Ba30.gydF4y2Ba].在具有密码子位置率异质性的严格时钟模型和一个大小恒定的合并树之前,系统发生的估计日期为1924年(95%最高后验密度(HPD): 1911 - 1936),该血清型的估计替代率估计为8.38 × 10gydF4y2Ba4gydF4y2Ba(95% hpd: 6.40 × 10gydF4y2Ba4gydF4y2Ba- 1.05 × 10gydF4y2Ba3gydF4y2Ba).gydF4y2Ba
总结系统发生树后验分布的一种方法是根据后验概率对树拓扑进行排序,并考虑最少代表的树的最小集合gydF4y2BaxgydF4y2Ba后验概率的%。这个集合被称为gydF4y2BaxgydF4y2Ba%可信树拓扑集合[gydF4y2Ba40gydF4y2Ba].出于假设检验的目的,如果在95%可信的树拓扑集中没有发现系统发育,则可以拒绝该系统发育。在这个例子中,我们发现对于不同的模型,可信集的大小有很大的不同。在表gydF4y2Ba1gydF4y2Ba我们列出了四个模型中每个模型的50%可信集大小的后验估计。我们选择50%是因为GTR + Γ + I模型都在分布的尾部采样了许多单例树,因此准确估计95%可信集的大小是不可行的。然而,该表清楚地表明,GTR + CP模型的后验分布几乎是一个数量级更集中在树空间。这表明,对于这个数据集,GTR模型既是一个更精确的估计器,也是一个更好的数据拟合器。在GTR + CP +严格模型的情况下,38的1.1919 × 10gydF4y2Ba17gydF4y2Ba在给定的数据下,有17个根结点的树占总概率的一半。gydF4y2Ba
结论gydF4y2Ba
BEAST是一个用于进化参数估计和假设检验的灵活分析包。BEAST中模型规范的基于组件的性质意味着可能存在的不同进化模型的数量非常大,因此很难进行总结。然而,BEAST软件的一些已发布的使用已经突出了该软件所享有的广泛应用[gydF4y2Ba6gydF4y2Ba,gydF4y2Ba8gydF4y2Ba,gydF4y2Ba34gydF4y2Ba,gydF4y2Ba37gydF4y2Ba,gydF4y2Ba41gydF4y2Ba].gydF4y2Ba
BEAST是一个积极开发的包,下一个版本的增强包括(1)树状的出生-死亡先验(2)更快、更灵活的基于密码子的替代模型(3)结构化合并,以模拟具有迁移的细分种群(4)连续特征进化模型和(5)基于随机局部分子钟的新的放松时钟模型。gydF4y2Ba
方法gydF4y2Ba
BEAST软件包的整体架构是一个文件中介的管道。核心程序以一个XML文件作为输入,该文件描述了要分析的数据、要使用的模型和MCMC算法的技术细节,如建议分布(操作符)、链长度和输出选项。BEAST分析的输出是一组由制表符分隔的纯文本文件,其中总结了估计的参数值和树的后验分布。gydF4y2Ba
一些额外的软件程序可以帮助生成输入和分析输出:gydF4y2Ba
•gydF4y2Ba美丽女孩gydF4y2Ba是一个用Java编写并随BEAST一起分发的软件包,它提供了一个图形用户界面,用于为许多简单的模型组合生成BEAST XML输入文件。gydF4y2Ba
•gydF4y2Ba示踪剂gydF4y2Ba是一个用Java编写的软件包,与BEAST分开发布,为MCMC输出分析提供图形化工具。它可以用于分析BEAST的输出以及其他常见MCMC包的输出,例如gydF4y2BaMrBayesgydF4y2Ba[gydF4y2Ba1gydF4y2Ba),gydF4y2BaBAli-PhygydF4y2Ba[gydF4y2Ba42gydF4y2Ba].gydF4y2Ba
由于BEAST XML输入格式的组合性质,不是所有模型都可以通过的图形界面指定gydF4y2Ba美丽女孩gydF4y2Ba.事实上,模型可能组合的绝对数量意味着,不可避免地,许多组合本质上是未经尝试和测试的。也有可能创建对正在分析的数据不合适或没有意义的模型。gydF4y2Ba美丽女孩gydF4y2Ba因此,目的是作为一种生成常用的和易于理解的分析的方法。对于更有冒险精神的研究人员,并记住上面的警告,可以直接编辑XML文件。有许多在线教程可以指导用户如何做到这一点。gydF4y2Ba
提供高度结构化的XML输入格式的主要动机之一是促进复杂演化分析的再现性。虽然交互式图形用户界面提供了令人愉快的用户体验,但对于用户来说,记录和再现所做选择的完整序列可能很耗时,而且容易出错,特别是在MCMC分析通常可用的大量选项的情况下。通过将图形用户界面(BEAUti)与分析(BEAST)分离,我们容纳了一个XML层,该层捕获正在执行的MCMC分析的确切细节。我们强烈建议在BEAST分析结果发布的同时,定期发布XML输入文件作为补充信息。由于MCMC分析的非平凡性质和提高再现性的需要,我们认为,发表任何贝叶斯MCMC分析的确切细节都应成为发表所有MCMC分析结果的先决条件。gydF4y2Ba
BEAST的输出是一种简单的以制表符分隔的纯文本文件格式,每个示例一行。当累积到频率分布时,该文件提供了每个参数(例如突变率、树高和种群大小等参数)的边际后验概率分布的估计。这可以使用任何标准的统计包,也可以使用专门编写的包,gydF4y2Ba示踪剂gydF4y2Ba[gydF4y2Ba43gydF4y2Ba].gydF4y2Ba示踪剂gydF4y2Ba提供了许多图形和统计方法来分析BEAST的输出,以检查性能和准确性。它还提供了专门的函数,用于在使用合并模型时总结种群大小随时间的后验分布。gydF4y2Ba
每个样本状态的系统发育树以NEWICK或NEXUS格式写入单独的文件。这可用于研究各种系统发育问题的后验概率,如某一特定生物群的单系性或获得一致的系统发育。gydF4y2Ba
尽管在程序的灵活性和计算性能之间总是存在权衡,但BEAST在大型分析(例如[gydF4y2Ba41gydF4y2Ba])。贝叶斯MCMC算法需要评估链中每个状态的可能性,因此性能取决于这些可能性评估的速度。BEAST试图通过仅重新计算模型中与前一状态相比发生变化的部分的可能性来最小化评估状态所花费的时间。在此基础上,用C语言实现了系统的核心计算功能。可以将其编译为针对给定平台的高度优化库,从而提高速度。如果没有找到这个库,BEAST将使用这些函数的Java版本,从而保持其平台独立性。gydF4y2Ba
参考文献gydF4y2Ba
Huelsenbeck JP, Ronquist F: MrBayes:系统发生树的贝叶斯推断。生物信息学,2001,17:754-755。10.1093 /生物信息学/ 17.8.754。gydF4y2Ba
博蒙特M:用微型卫星探测人口的扩张和衰退。中国生物医学工程学报,1999,29(3):344 - 344。gydF4y2Ba
Drummond AJ, Nicholls G, Rodrigo A, Solomon W:从时间间隔序列数据同时估计突变参数,群体历史和系谱。中国生物医学工程学报,2002,26(3):357 - 357。gydF4y2Ba
Wilson I, Weale M, Balding D:从DNA数据推断:种群历史,进化过程和法医匹配概率。《中国社会统计》,2003,26(1):1 - 5。10.1111 / 1467 - 985 x.00264。gydF4y2Ba
杨震,杨晓明,杨晓明:基于贝叶斯估计的物种分化时间和祖先群体大小。遗传学报,2003,34(3):344 - 344。gydF4y2Ba
Pybus O, Drummond AJ, Nakano T, Robertson B, Rambaut A:埃及丙型肝炎病毒的流行病学和医源性传播:贝叶斯联合方法。中华生物医学杂志,2003,20:381-387。10.1093 / molbev / msg043。gydF4y2Ba
库纳M: LAMARC 2.0:种群参数的最大似然和贝叶斯估计。生物信息学,2006,22:768-770。10.1093 /生物信息学/ btk051。gydF4y2Ba
王晓明,王晓明,王晓明,王晓明,王晓明。基于贝叶斯共估计方法的植物系统发育研究。中国生物医学工程学报,2005,26(3):344 - 344。gydF4y2Ba
Redelings B, Suchard M:对齐和系统发育的联合贝叶斯估计。中国生物工程学报,2005,34(4):344 - 344。10.1080 / 10635150590947041。gydF4y2Ba
大都会N, Rosenbluth A, Rosenbluth M, Teller A, Teller E:通过快速计算机计算状态方程。化学与物理学报,2003,21:1087-1092。10.1063/1.1699114。gydF4y2Ba
马可夫链蒙特卡罗抽样方法及其应用。生物计量学学报,2000,27(3):377 - 377。10.1093 / biomet / 57.1.97。gydF4y2Ba
朱克曼坎德,鲍林,李:蛋白质的进化分化和趋同。1965年,纽约:学术出版社,97-166。gydF4y2Ba
Sanderson M:在没有速率常数的情况下估计发散时间的非参数方法。分子生物学与进化。1997,14:1218-1231。gydF4y2Ba
索恩J,岸野H,画家I:估计分子进化速率的进化速率。分子生物学与进化,1998,15:1647-1657。gydF4y2Ba
Rambaut A, Bromham L:从分子序列估计散度日期。分子生物学与进化,1998,15:442-448。gydF4y2Ba
杨忠,杨志刚:基于局部分子钟的灵长类物种形成时间估算。分子生物学与进化,2000,17:1081-1090。gydF4y2Ba
Kishino H, Thorne J, Bruno W:概率模型下的发散时间估计方法的性能。分子生物学与进化,2001,18:352-361。gydF4y2Ba
Sanderson M:估计分子进化和分化时间的绝对速率:惩罚似然方法。分子生物学与进化,2002,19:101-109。gydF4y2Ba
孙晓东,王晓明,王晓明。基于多位点数据的进化速率估计。中国生物医学工程学报,2002,29(4):529 - 529。10.1080 / 10635150290102456。gydF4y2Ba
Aris-Brosou S, Yang Z:情景演化的贝叶斯模型支持前寒武纪晚期后生动物的爆发性多样化。中国生物医学工程学报,2004,19(4):344 - 344。10.1093 / molbev / msg226。gydF4y2Ba
Drummond AJ, Ho S, Phillips M, Rambaut A:轻松的系统发育和自信的年代测定。《科学公共图书馆·生物学》,2006,4:e88-10.1371/journal.pbio.0040088。gydF4y2Ba
Lanave C, prepareata G, Saccone C, Serio G:一种计算进化替代率的新方法。分子进化学报,1984,20:86-93。10.1007 / BF02101990。gydF4y2Ba
DNA序列分析中的一些概率和统计问题。数学与生命科学,1986,17:57-86。gydF4y2Ba
长谷川M, Kishino H, Yano T:用线粒体DNA分子钟测定人猿分裂。分子进化学报,1985,22:160-174。10.1007 / BF02101694。gydF4y2Ba
杨智,杨志勇:基于密码子的蛋白质编码DNA序列的核苷酸替代模型。分子生物学与进化,1994,11:725-736。gydF4y2Ba
Uzzell T, Corbin K:拟合进化事件的离散概率分布。科学通报,1997,33(4):379 - 379。10.1126 / science.172.3988.1089。gydF4y2Ba
杨智:基于可变频率DNA序列的最大似然系统发育估计:近似方法。中国生物工程学报,2004,26(3):357 - 357。10.1007 / BF00160154。gydF4y2Ba
顾旭,傅艳,李伟:核苷酸位点替代率异质性的极大似然估计。分子生物学与进化。1995,12:546-557。gydF4y2Ba
Waddell P, Steel M:一般时间可逆距离与不等速率跨站点:混合伽玛和逆高斯分布不变的站点。分子系统发育与进化,1997,8:398-414。10.1006 / mpev.1997.0452。gydF4y2Ba
Rambaut A:估计分子进化的速度:将非同期序列纳入最大似然系统发育。生物信息学,2000,16:395-399。10.1093 /生物信息学/ 16.4.395。gydF4y2Ba
Drummond AJ, Pybus O, Rambaut A, Forsberg R, Rodrigo A:可测量进化的种群。生态学报,2003,18:481-488。10.1016 / s0169 - 5347(03) 00216 - 7。gydF4y2Ba
Kingman J:凝聚剂。随机过程及其应用。1982,13(3):366 - 366。10.1016 / 0304 - 4149(82) 90011 - 4。gydF4y2Ba
葛瑞芬,李志刚,李志刚:不同环境下中性等位基因的抽样理论。中国生物医学工程学报,2004,27(4):344 - 344。10.1098 / rstb.1994.0079。gydF4y2Ba
杨晓明,王晓明,杨晓明。基于分子序列的种群动态贝叶斯聚结推理。分子生物学与进化,2005,22:1185-1192。10.1093 / molbev / msi103。gydF4y2Ba
威尔逊A,萨里奇V:人类进化的分子时间尺度。美国国家科学研究院。1969, 63: 1088-1093。10.1073 / pnas.63.4.1088。gydF4y2Ba
杨晓东,王晓明,王晓明。DNA序列最大似然排列的进化模型。分子进化学报,1997,33(4):344 - 344。10.1007 / BF02193625。gydF4y2Ba
李志刚,李志刚,李志刚,李志刚。HIV-1群体的分子群体遗传学研究进展。中国生物医学工程学报,2004,27(2):357 - 357。10.1534 / genetics.104.026666。gydF4y2Ba
牛顿M, Raftery A:加权似然自举近似贝叶斯推理。皇家统计学会学报,1994,56:3-48。gydF4y2Ba
夏皮罗B, Rambaut A, Drummond AJ:选择合适的替代模型用于蛋白质编码序列的系统发育分析。分子生物学杂志,2006,23:7-9。10.1093 / molbev / msj021。gydF4y2Ba
胡森贝克J, Rannala B:系统发生树在简单和复杂替代模型下的贝叶斯后概率的频率特性。中国生物工程学报,2004,29(4):344 - 344。10.1080 / 10635150490522629。gydF4y2Ba
Shapiro B, Drummond AJ, Rambaut A, Wilson MC, Matheus PE, Sher AV, Pybus OG, Gilbert MTP, Barnes I, Binladen J, Willerslev E, Hansen AJ, Baryshnikov GF, Burns JA, Davydov S, Driver JC, Froese DG, Harington CR, Keddie G, Kosintsev P, Kunz ML, Martin LD, Stephenson RO, Storer J, Tedford R, Zimov S, Cooper A:白林琴草原野牛的兴衰。科学通报,2004,30(3):344 - 344。10.1126 / science.1101074。gydF4y2Ba
Suchard M, Redelings B: BAli-Phy:同步贝叶斯推理的对齐和系统发育。生物信息学,2006,22:2047-2048。10.1093 /生物信息学/ btl175。gydF4y2Ba
Rambaut A, Drummond AJ: Tracer[计算机程序]。2003年,(gydF4y2Bahttp://beast.bio.ed.ac.uk/tracergydF4y2Ba]gydF4y2Ba
确认gydF4y2Ba
我们要感谢罗尔德·福斯伯格,约瑟夫·赫里德,菲利普·莱梅,格顿·伦特,西德尼·马科维茨,奥利弗·皮布斯,贝斯·夏皮罗,科尔比尼·斯特里默和马克·苏查德的宝贵贡献。AJD得到了威康基金会的部分支持,AR得到了英国皇家学会的支持。gydF4y2Ba
作者信息gydF4y2Ba
作者及隶属关系gydF4y2Ba
相应的作者gydF4y2Ba
额外的信息gydF4y2Ba
作者的贡献gydF4y2Ba
在2002年6月至2007年10月期间,AJD和AR设计并实现了BEAST的所有版本(版本1.4.6)。BEAST源代码的一部分是基于AJD(称为MEPI)在2000年至2002年期间在奥克兰大学攻读博士学位期间开发的原始马尔科夫链蒙特卡罗程序。BEAST源代码的一部分是基于AR以前开发的c++软件。两位作者都对本文的写作做出了贡献。gydF4y2Ba
电子辅助材料gydF4y2Ba
12862 _2007_505_moesm1_esm.xmlgydF4y2Ba
附加文件1:Dengue4-GTR-CP-strict。对BEAST输入的XML文件进行GTR + CP +严格的时钟分析。(xml37 kb)gydF4y2Ba
12862 _2007_505_moesm2_esm.xmlgydF4y2Ba
附加文件2:dengue4 - gtr - cp -relax。野兽输入XML文件进行GTR + CP +放松时钟分析。(xml38kb)gydF4y2Ba
12862 _2007_505_moesm3_esm.xmlgydF4y2Ba
附加文件3:Dengue4-GTR-GI-strict。对BEAST输入的XML文件进行GTR + Γ + I +严格的时钟分析。(xml35kb)gydF4y2Ba
12862 _2007_505_moesm4_esm.xmlgydF4y2Ba
附加文件4:dengue4 - gtr - gi -relax。野兽输入的XML文件为GTR + Γ + I +放松时钟分析。(xml36kb)gydF4y2Ba
作者提交的图片原始文件gydF4y2Ba
下面是作者提交的原始图片文件的链接。gydF4y2Ba
权利和权限gydF4y2Ba
本文由BioMed Central Ltd授权发布。这是一篇开放获取文章,根据创作共用属性许可协议(gydF4y2Bahttp://creativecommons.org/licenses/by/2.0gydF4y2Ba),允许在任何媒介上不受限制地使用、传播和复制,前提是正确地引用原始作品。gydF4y2Ba
关于本文gydF4y2Ba
引用本文gydF4y2Ba
德拉蒙德,a.j.,兰姆波特,a.b. BEAST:抽样树的贝叶斯进化分析。gydF4y2BaBMC Evol BiolgydF4y2Ba7gydF4y2Ba, 214(2007)。https://doi.org/10.1186/1471-2148-7-214gydF4y2Ba
收到了gydF4y2Ba:gydF4y2Ba
接受gydF4y2Ba:gydF4y2Ba
发表gydF4y2Ba:gydF4y2Ba
DOIgydF4y2Ba:gydF4y2Bahttps://doi.org/10.1186/1471-2148-7-214gydF4y2Ba
关键字gydF4y2Ba
- 马尔科夫链蒙特卡洛gydF4y2Ba
- 边际似然gydF4y2Ba
- 马尔科夫链蒙特卡罗算法gydF4y2Ba
- 高后验密度gydF4y2Ba
- 可逆跳跃马尔可夫链蒙特卡洛gydF4y2Ba