文摘
背景
阿勒格尼woodrats (Neotoma高地”)被发现在整个室内阿巴拉契亚高地和异质种群分布。历史上这些异质种群持续相对流体网络,使亚种群间基因流和开拓殖民地以前报告的地区。然而,在过去的45年,丰富的阿勒格尼woodrats拒绝整个物种的范围由于栖息地的破坏,减少硬桅杆的可用性,和蛔虫寄生。为了启动基因拯救小,遗传发育不全的族群在新泽西州,woodrats是从基因易位健壮的人口在宾夕法尼亚州(PA)在2015年,2016年和2017年。在此,我们评估这些易位恢复的疗效在收件人人口遗传多样性。
结果
我们设计了一个新颖的134个单核苷酸多态性面板,用来基因型六woodrats转移从新泽西PA和82人人口捕获的易位前后事件。这些数据表明,至少两个转移个人成功地产生了至少13的后代,他们复制。此外,全民观察第一组易位后大幅上涨,达到的水平与人口在印第安纳州和俄亥俄州,在随后几年,一直升高。丰度也增加了监测期间,暗示宾夕法尼亚易位启动基因新泽西人口的救助。
结论
令人鼓舞的是,我们的研究结果表明非常小数量的易位个人可以成功恢复威胁人口的遗传多样性。我们的工作也强调了管理的挑战非常小的种群,比如当转移个人有更大的繁殖成功率相对于居民。最后,我们注意到正在进行的工作和阿勒格尼woodrats可能广泛地塑造我们理解异质种群内的遗传救援和跨异构的风景。
背景
景观尺度人为干扰会导致栖息地的丧失和碎片,因此空间隔离当地野生动物种群和阻碍功能连通性1,2,3,4]。物种通常结构异质种群可能特别受到空间隔离种群的威胁。当地消灭补丁可以共同和持久性的metapopulation取决于正在进行的开拓殖民地事件(5,6,7]。因此,中断传播和基因流栖息地站点可以降低全民遗传变异性和健身,促进自然消灭小亚种群,防止再度移民事件,威胁metapopulation持久性(4,5,8,9]。阿勒格尼woodrats (Neotoma高地”)需要摇滚栖息地(如悬崖,距骨山坡,博尔德字段),位于高海拔地区主要在阿巴拉契亚山脉和内陆高地(10,11]。这些网站是自然分离的栖息地。因此,woodrats通常形成小亚种群(定义为合适的栖息地岩石),由分散在一个更大的metapopulation[相连12,13]。如果运动生境斑块之间中断,亚种群变得孤立,基因流动受到抑制时,遗传多样性丢失通过漂移,近交衰退发生时,种群数量减少,开拓殖民地报告网站的下降(14,15,16]。
新泽西州(NJ)是一个残余阿勒格尼woodrats人口,位于~ 240公里距离最近的现存人口在宾夕法尼亚州。个体采样2009年、2011年和2012年和基因分型在11个微卫星位点的遗传变异相对较低,所表示的等位基因多样性和观察到的杂合性(附加文件1)。为了保护担忧与遗传多样性下降,新泽西DEP鱼类和野生动物介绍六个人基因强劲的人口2015年在宾夕法尼亚州,2016年和2017年的假设下,如果转移个人复制,人口数量和遗传变异会增加(即。、遗传的解救;(17,18,19,20.])。然而,确定证据的繁殖成功率和量化遗传变异性依赖于识别标记面板与合适的统计力量(21,22,23]。
板的单核苷酸多态性(snp)可以产生一个低概率的身份(PID)(即。,the likelihood that two randomly chosen individuals in a population will present seemingly identical genotypes; [21]),从而帮助准确重建家族关系(24,25,26),即使人口是天生的23]。甚至相对较小的SNP面板(例如,58 - 109标记)最终可以执行及微卫星(或比小套房23,27,28,29日,30.]。为此,我们阿勒格尼woodrat基因组测序和注释草案基因组组装。我们随后设计了134个SNP面板结合了相关的基因和推定地中性标记。我们进行了初步分析,探讨SNP检测是否为个人提供了更大的统计力量比常用的微卫星标记面板标识。SNP位点被用来评估遗传多样性的变化后易位到新泽西的剩余人口和识别转移人的后代。
结果
核基因组测序和SNPtype试验发展
我们生成137.6 Gb (Gb)的原始序列数据n高地”,包括从paired-end 119.8 Gb (PE)图书馆和17.8 Gb的mate-paired (MP)图书馆(附加文件2)。我们的核基因组组装草案包括60789支架大于2000完全(bp)的长度。我们使用车身v5评估基因组的完整性通过识别mammalia_odb10直接同源,发现77.9%的万能单副本直接同源是完整的(77.2%的单一副本,0.7%的复制),8.7%的支离破碎和13.4%的失踪。
我们最初确定的627421年高质量的单核苷酸多态性。这些,我们选择了192个snp在Fluidigm包括SNPtype化验。我们随后排除58位点原因中概述的方法(例如,数据没有集群独特的纯合子和杂合的状态)。剩余的134个位点,至少128位点放大为每个318 woodrats基因分型(附加文件3)。这些位点可以大致分为相关的基因(n = 72)和中性标记(n = 62)。
使用微卫星和SNP标记概率的身份
50型woodrats捕获亚当斯县在2017年和2019年,俄亥俄州(OH)在11个微卫星标记导致PID4.0×105和兄弟姐妹之间的概率的身份(PIDsib) 9.8×103。相比之下,134个SNP位点产生5.0×10的值-27年(PID)和3.1×10-14年(PIDsib)。如果70位点的更为保守的数据集,PID是1.9×10-13年PIDsib是3.1×107在所有50个人。此外,我们的研究结果表明,一个小得多的snp可能用于后续研究小组实现PID< 0.0001(附加文件4;一个PID < 0.0001被认为是足够低区分甚至在最自然的人口(个人密切相关31日- - - - - -33])。鉴于这些结果,所有其他样本基因分型用苏格兰民族党面板。值得注意的是,有一个显著的、积极的关系杂合的微卫星位点的数量每个体和杂合的SNP位点的数量(线性回归:r2= 0.32,p < 0.0001,额外的文件5)。
遗传变异性和生殖成功后易位栅栏人口
血统分析显示,六个woodrats转移从宾夕法尼亚州到新泽西产生最少的(表13的后代1)。女性内进行2015年生产的至少有四个孩子在2016年和2019年之间。2015年男性内进行生产至少有九个孩子,主要是在2016年(表1)。2015男女易位产生的后代至少13个孩子自己的2017年和2019年之间(表1)。我们发现没有证据表明从捕获和随后的基因分型,其他四个内进行个人复制。雄性的转移在2016年和2017年被证实死亡11和1周内发布,分别。摄像头捕获视频的女性发生易位与一只小狗在2016年。目前尚不清楚这只小狗成年前死亡或者只是避免陷阱,是拍摄的位置是定期捕获以外的区域。女2017年发生易位也解决了常规之外的捕获区域并没有检测到。
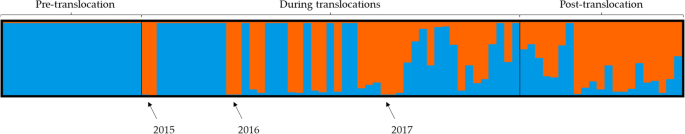
的82人口从栅栏组织样本收集,收集五个2009年,2011年十三,2015年九,2016年十八岁,16 2017年,8 2018年和2019年13。一次Bonferroni调整应用,单个位点被发现的哈迪温伯格平衡,且仅在2019年(展示杂合子的证据)。结构分析居民个人和那些转移到新泽西的证据两个遗传学上截然不同的集群提供当最保守的数据集(70位点)是利用。全民的遗传成分改变了2015年易位事件后,作为从蓝色转变说明了集群与常住人口相关人工中介基因流之前,橙色的增加集群与PA个体的遗传资料(无花果。1)。尽管如此,所有等位基因历史上出席的位点被认为是本研究后保留易位(数据没有显示)。
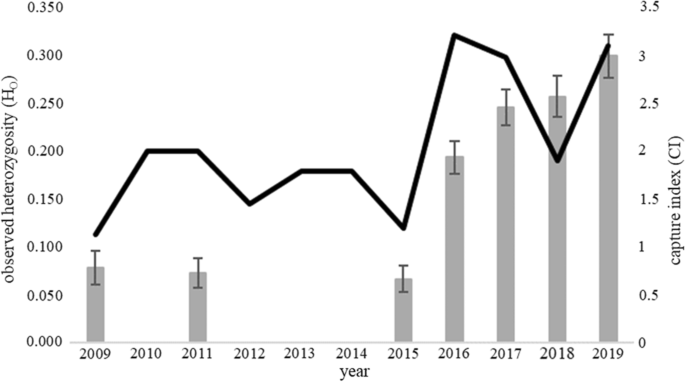
易位之前,观察到的杂合性(HO)和预期的杂合性(HE在新泽西)显著低于在网站在印第安纳州和俄亥俄州(表2)。然而,遗传变异在新泽西人口的增长特别是易位(表之后2、表3,无花果。2)。例如,观察杂合度增加从2009年的0.08±0.02,0.30±0.02 2019年(表3,无花果。2)。平均HO和HE可比在印第安纳州,俄亥俄州和post-translocation新泽西(表吗2,3,无花果。2)。
我们发现20出版物的Fluidigm SNPtype试验被用来在相对较少的位点基因型个体(38 - 192个snp)和HO和/或HE据报道(表4)。物种的成员描述辐鳍鱼纲,鸟纲,双壳纲,哺乳类和主要认为是世界自然保护联盟的“最少关注”。在研究中,HO和HE范围从0.13到0.45和0.14 - 0.42;分别(表4)。绝大多数的HO和HE估计物种特征为“最少关注”(除了两个)下跌0.25和0.37之间。中值HO和HE分别为0.32和0.31。
讨论
基因组测序和SNP分析发展
所述数据仅代表第二次属的一个成员Neotoma经历了全基因组测序(34]。遗传资源Neotoma高地”尤其有限(35),然而,即使是低覆盖率测序可用于生成工具,通知管理濒危物种。例如,两个车道的paired-end测序和启用一巷mate-paired测序组装完整的线粒体基因组的35)和134个SNP位点的识别描述在这个手稿。研究表明,在某些情况下SNP基因分型能更好地揭示人口结构精细,提供证据的微分选择在人口比其他标志板,宽估计基因杂合性(36,37,38,39,40,44]。50型woodrats使用微卫星和SNP位点表明我们的SNP分析提供了分析统计能力增强。此外,PIDsib估计也很低,表明该小组可以用来区分woodrats即使相关个人存在于人口(31日- - - - - -33]。考虑到许多woodrat空间隔离的人群(例如,残余NJ栅栏人口),存在个人应该假设密切相关。最后,DNA提取自然脱落的羽毛、头发和粪便样本和随后Fluidigm SNP基因分型结果被用来确定个人金雕[24),狼,野猫和熊32,33]。考虑到低PID估计与此相关分析,我们预计类似的方法可用于无创性监测阿勒格尼woodrat人群头发或粪便样本。
时间变化的遗传变异性易位到新泽西的剩余人口
保护经理一直担心易位在扩展地理距离会导致相对更大的介绍和居民个人之间遗传距离,增加远系繁殖抑郁的风险(45]。然而,最近的研究表明,异族通婚抑郁症很少有负面影响易位的成功项目(19,46]。此外,等因素改变个体的遗传多样性可能是更好的预测健身后介绍小说的人口比遗传距离(47]。尽管相对的地理来源之间的距离和居民人口固有在这项研究中,成功的繁殖进行个人显然开车增加在随后几年的遗传变异性。血统分析提供了证据表明,至少有两个woodrats转移从2015年宾夕法尼亚州新泽西继续繁殖,他们的后代也是如此。增加HO和HE明显的只要2016,使观察到的水平与woodrat人群的哦,和坚持到年底监测期在2019年。遗传变异性相比我们还在新泽西人口的其他物种和确定观察到新泽西的杂合性woodrats抓住2015年之前明显低于任何物种由世界自然保护联盟列为“最少关注”。易位后,HO和HE新泽西人口属于跨物种生成的范围的估计。增加数量自2015年以来提供了额外的证据的潜在基因救援。这样,本研究加入提供相对较少的证据增加人口规模或增长率后协助基因流(了19])。
管理的影响
正在进行的研究保护阿勒格尼woodrats可能通知把个人非常小的人群的最佳实践。管理指南建议把一些非居民个人,代表接收方20%的人口减少的可能性淹没了当地自适应遗传变异(17,20.]。基因(即被淹。,the rapid increase in frequency of alleles introduced by gene flow; [48,49])可能导致的损失私有等位基因在收件人人口。反过来,这可能导致的损失species-wide等位基因多样性(49),甚至常住人口的遗传多样性增加。努力减少遗传淹没会导致把很少的人当收件人种群丰度较低。本研究加入其他暗示成功繁殖只有一到三个非居民个人可以促进增加遗传多样性和丰富50,51,52,53]。在某些情况下,然而,这些移民实现大幅提高繁殖成功率相比,居民个人,导致近亲繁殖的后代(如北极狐,[51];狼,54,55,56])。即使没有直接观察的近亲繁殖,繁殖斜已知有效的人口规模和减少导致加速遗传多样性的损失由于漂移57]。不成比例的繁殖成功率进行个人可能是常见的阿勒格尼woodrats如果人口性别比例失衡在小50),居民个人更倾向于与转移个人作为避免近亲繁殖交配机制(58),或F1后代增加了健身源于杂种优势(59,60,61年]。这项研究和戴维斯et al。(5039]文档的观察进行男阿勒格尼woodrats为了繁衍和35%的年轻人被困在随后的赛季,在各自的人口。几个已知实例的近亲繁殖在亲属后代(表中2,(50)但是,令人鼓舞的是,与此同时稳定或增加全民遗传变异性和丰富。保护管理者监控小种群可以考虑基因型个体,进行亲子关系分析、遗传多样性和监测年度或每年。这将允许快速易位的额外个人小人口如果提高繁殖成功率似乎导致近亲繁殖活动,或者,补充之前,在遗传救援失败(即。如果非居民生存很低)。值得注意的是,即使没有近亲繁殖,真正孤立的人群(如栅栏的)最终将需要额外的人工中介基因流来抵消损失由于遗传漂变的遗传多样性。
还有其他的方法进行的研究woodrat易位有潜力增加深度对我们理解遗传救援和恢复。遗传救援的研究通常认为人口作为离散单元,不间断的景观功能。woodrats存在的自然倾向异质种群为科学家提供的机会来研究基因在异构的风景和救援,特别是,如何等位基因引入一个栖息地补丁可能在移动网站。正如最近的研究提出了选择特定个人的能力减少基因近交衰退萎缩的人口(19,62年),特定栖息地站点可能针对版本如果他们自然传播通道连接到metapopulation的其他部分。的确,最近的工作在景观遗传学弗吉尼亚的阿勒格尼woodrats表明,海拔低,而不是人为的障碍,如道路,可能阻止改变个人和/或他们的后代分散在栖息地的网站(63年]。
结论
在此,我们描述一个新颖的SNP分析,它提供了增加统计研究的一种常见小和血缘的人群。我们的研究有着重要的意义,可以让残留种群的濒危物种的地理距离最近的metapopulation孤立。把少量个体非常小的数量可能会增加风险的生殖斜其次是遗传漂变和近亲繁殖,必须增加监测以下介绍。尽管如此,人工中介基因流动可能是不可或缺的残留种群的持久性。我们的结果表明,令人鼓舞的是,少量的引入,基因变量个体能够成功繁殖,增加全民遗传多样性,促进大量增加。
方法
基因组组装和注释
我们提取的脱氧核糖核酸(DNA)从单一的尾巴夹n高地”个人通过配对商用提取(DNEasy血液和组织、试剂盒、Venlo,荷兰)和清理(DNA清洁&集中器,Zymo研究,欧文,加利福尼亚州)在符合制造商的指示。我们进行了三车道paired-end和mate-paired测序使用Illumina公司HiSeq2500巷之一。我们使用Trimmomatic [64年]删除适配器,丢弃短读和修剪质量差基地从5′和3′末端的原始序列读取所述斯科菲尔德et al ., (35]。我们使用深渊1.9.0 [65年)进行几个组件从40到85公里长度。PE读取被用来产生重叠群。议员读取被用来推断出秩序,方向,和重叠群之间的距离,在支架连接在一起。最长最大的装配将军曾经价值和支架用于下游分析。车身v5 (66年),由gVolante实现(67年),被用来评估基因组的完整性。
我们使用了制造商2.28管道(68年)注释所有大于10 kb的支架,后道尔描述的方法等。69年和柯南道尔et al。26]。简要总结,我们第一次使用Repeat-Masker识别和面具的重复DNA。第二,我们下载6762亩骶从UniProtKB蛋白质序列数据库(www.uniprot.org)和protein2genome设置用于制造商产生基因注释。这些注释随后被用来训练吸附(70年)并生成从头开始预测。第三,我们一致的蛋白质序列和93400年亩骶表达序列标签(EST)的基因组序列使用爆炸和InterProScan用来识别领域公认的蛋白质。最后,所有从头开始由蛋白质基因预测,美国东部时间或者InterProScan证据被提升为基因注释。
SNP发现和分析设计
我们确定了snp在柯南道尔et al。26]。简而言之,我们一致paired-end读回基因组组装使用草案BWA 0.7.12 [71年2.3)和使用皮卡德(http://broadinstitute.github.io/picard)分类和识别重复的读取。我们使用了GATK 3.6管道(72年,73年]在indels调整读取和识别高质量的单核苷酸多态性与phr质量分数≥30。然后我们选择95年常染色体核标记与基因沙漠(即有关。,“neutral” markers) and 97 autosomal nuclear markers associated with protein-coding genes. We deliberately chose no more than one SNP of each category from a given scaffold to minimize linkage disequilibrium. To identify neutral markers, we quantified the distances between all SNPs and genes using the BEDtools suite [74年),最终选择标记基因的95%百分位距离。我们使用SnpEff 4.3 [75年)找到与非同义单核苷酸多态性相关基因的改变其实区域(即。“相关的基因标记)。进口2.3 [76年)被用来确认至少60高质量的旁侧序列的核苷酸在场上游和下游的标记,这guanine-cytosine (GC)含量小于65%,而且没有其他变量网站在20核苷酸在场。
DNA提取和SNP基因分型
我们被困,随后82组woodrats抽样从栅栏,新泽西州2009年和2019年之间(表2,3),包括18和64个人采样前后易位开始,分别。我们遵循标准live-trapping协议(77年,78年),收集了2毫米从每个耳朵穿孔,这是保存在70 - 100%的乙醇。每年捕获发生,我们计算一个捕捉索引数除以独特的个体被陷阱晚上和乘以10的数量(79年]。计算一个陷阱索引允许我们控制夜晚捕捉的数量差异发生在年77年]。使用醋酸铵提取DNA进行协议(14)或Zymo快速dna Miniprep +工具包。我们使用Fluidigm®BioMark HD型™系统这些个体基因型。此外,我们六个人的基因转移从宾夕法尼亚州到新泽西在2015和2017之间。主义式的最后,我们172和58样本收集来自印第安纳州和俄亥俄州,分别(表2)。这些样本收集2015年至2019年长期监测研究的一部分。
SNP调用编辑使用Fluidigm®的基因分析软件。我们排除了标记从下游分析数据时没有集群成不同的纯合子和杂合的状态,如果小等位基因频率小于0.025。我们使用卡方测试由GenAlEx实现(24,26)为偏离哈迪温伯格平衡测试。Bonferroni调整后,单个位点被发现一致的跨年,哈迪温伯格平衡问题却被忽略了,留下134位点。我们排除了个人分析如果≥7位点没有成功的基因,如话费与基因分型错误往往是负相关24,32]。使用snpStats [80年),我们发现了一个数量的标记连锁不平衡,但认为,在很多情况下这是由于血缘关系,而不是两个标记在基因组接近。然而,为了满足Cervus 3.0.7的假设(81年2.3.4)和结构(82年,83年),我们确认所有成对比较r2> 0.2和删除一个标记在每种情况下,创建一个减少数据集的70位点snp都在连杆平衡。
使用微卫星和SNP标记概率的身份
先前的研究的n高地”利用时间为相对较小的电池板月11日至微卫星标记(例如,15,63年,84年,85年])。评估与每种方法相关的统计力量,我们组50 woodrats亚当斯在2017年和2019年在县哦11微卫星和134个SNP位点(附加文件3和额外的文件4)。随后我们计算的概率随机选择两个个体的人口将有相同的基因型(PID),使用每一个标记面板。我们另外PIDsib计算,它代表了一种保守的上界的可能性两个个体采样偶然从一个人口将有相同的基因型(32,33]。这估计是特别有用,当子结构存在于人口(即。相关的个人;(32,33])。
繁殖成功率和遗传变异性易位栅栏
我们使用Cervus 3.0.7 [81年)分配个体采样大坝和雄在2015年和2019年之间。首次为个人被困在每年,所有woodrats困在同年和前几年被认为是候选人的父母。模拟包括100000复制周期。候选人大坝和雄抽样的比例估计为0.80,根据可比捕获捕获的概率估计方法的其他woodrat人口(16]。输入位点的比例是0.99和位点的比例出现打印错误设置为0.04 (26]。最低血统赋值的置信水平为95%。
2.3.4结构(82年,83年)、结构收割机0.6.94 [86年],Clumpak [87年)被用来可视化外加剂在栅栏,NJ woodrat人口跨越时间。我们利用减少数据集(即。,with all 70 loci in linkage equilibrium, see above) for 82 individuals sampled between 2009 and 2019, as well as the six individuals translocated from PA. We considered values of K = 1–8, running each value 10 times with an initial burn-in of 100,000 Markov chain Monte Carlo (MCMC) iterations and 1,000,000 subsequent iterations for each value. We assumed an admixture ancestry model and allowed for correlated allele frequencies [82年]。结果解释使用意味着可能性的K值和ΔK [86年]。
我们使用GenAlEx [88年)计算等位基因频率和预期,年前观察杂合度(2009、2011),(2015 - 2017)期间和之后易位(2018、2019)栅栏,新泽西人口。为我们提供上下文的解释时间栅栏人群的遗传变异性的改变,我们(1)使用GenAlEx [88年]计算等位基因频率和预期,观察到的杂合性在印第安纳州,新泽西和俄亥俄州和文献调查(2)观察和预期估计的杂合性生成使用Fluidigm®BioMark HD型™系统和相对较小的SNP检测(如96 - 192位点)。进行文献综述,我们搜索的短语“SNP类型分析”,“SNPtype试验”,“Fluidigm SNP分析”、“Fluidigm SNP芯片”和“基因分型结果Fluidigm分析软件”在谷歌学者。所有研究的非人类动物观察和/或预期的杂合性描述,我们记录的位点分析,样本大小,指标的遗传变异性及国际自然保护联盟(IUCN)的地位。如果平均HO和HE没有提供,我们平均每个值或特定人群值时。当解释这些结果,重要的一点是,我们没有一个健壮的了解SNP的HEHO和等位基因多样性随体型等分类,保护状态,栖息地,迁徙行为,分类组和营养类。相比之下,这些关系都进行了广泛的研究利用微卫星位点的遗传变异生成估计(89年,90年,91年,92年,93年]。
可用性的数据和材料
生成的数据集和/或分析在当前的研究中可在NCBI的短阅读档案(BioSample加入# SAMN25554335, BioProject加入# PRJNA802531),森林女神(https://doi.org/10.5061/dryad.rjdfn2zdb与此相关的手稿)和补充文件。
缩写
- NJ:
-
新泽西
- 单核苷酸多态性:
-
单核苷酸多态性
- Gb:
-
碱基
- 英国石油公司:
-
完全
- kb:
-
个碱基
- 体育:
-
Paired-end
- 议员:
-
Mate-paired
- 哦:
-
俄亥俄州
- PID:
-
概率的身份
- PIDsib:
-
兄弟姐妹之间身份的概率
- PA:
-
宾西法尼亚
- HO:
-
观察到的杂合性
- HE:
-
预期的杂合性
- :
-
印第安纳州
- 背景:
-
脱氧核糖核酸
- 美国东部时间:
-
表达序列标签
- GC:
-
Guanine-cytosine
- 密度:
-
马尔可夫链蒙特卡罗迭代
- 世界自然保护联盟:
-
国际自然保护联盟
引用
Wilcox BA、墨菲弟弟。保护策略:碎片在灭绝的影响。Nat。1985; 125:879 - 87。
年轻的一个,博伊尔T,布朗T .人口基因对植物生境破碎的后果。趋势生态另一个星球。1996;11:413-8。
Griffiths MikolašM, Tejkal M, Kuemmerle T, P, Svoboda M, Hlasny T, et al。森林管理影响北欧雷鸟(Tetrao urogallus)栖息地分布在喀尔巴阡山和连通性。Landsc生态。2017;32:163 - 79。
布兰顿再保险公司Cashner MF,托马斯先生,布兰德SL,弗洛伊德。栖息地的分裂导致增加之间的隔离和低濒危的种群内遗传多样性肯塔基箭头镖鲈(Etheostoma矢耳石spilotum)。Conserv麝猫。2019;20:1009-22。
Saccheri我Kuussaari M Kankare M, Vikman P, Fortelius W, Hanski即近亲繁殖和一只蝴蝶metapopulation灭绝。大自然。1998;392:491-4。
Hanski i Metapopulation生态。牛津:牛津大学出版社;1999年。
达拉斯助教,Saastamoinen M,舒尔茨T, Ovaskainen o .的地方和区域的相对重要性metapopulation动力学过程。J动物生态。2020;89:884 - 96。
Couvet d .有害的影响限制分散的人群的基因流动。Conserv杂志。2002;16:369 - 76。
野中郁次郎E,警笛J, Somervuo P, Ruokolainen L, Ovaskainen O, Hanski即扩大近交衰退从个人异质种群的影响。J动物生态。2019;88:1202-14。
普尔素描的阿勒格尼woodrat大肠的生活历史。J哺乳动物。1940;21:249 - 70。
Castleberry某人Mengak MT,福特WM。Neotoma高地”。Mamm物种。2006;789:1-5。
Hassinger J, Butchkoski C, Diefenbach d表面岩石社区管理Neotoma高地”。莱特:贝利J, J,编辑。阿勒格尼Woodrat:生态、减少物种的保护和管理。纽约:施普林格;2008年。133 - 52页。
木头p Woodrat种群动态和运动模式。莱特:贝利J, J,编辑。阿勒格尼Woodrat:生态、减少物种的保护和管理。纽约:春天出版公司;2008年。45 - 62页。
smyster TJ,杜尚我,约翰逊SA,拉金莱托,罗兹OE。metapopulation崩溃的后果:比较两个阿勒格尼之间的基因属性woodrat异质种群。Conserv麝猫。2012;13:849-58。
smyster TJ,页面路,大学W,尤金·O Jr。协同压力和困境的保护在一个多元的世界:一个案例研究在阿勒格尼woodrats。动画Conserv。2012; 15:205-13。
smyster TJ, Stauffer通用电气,约翰逊SA,哈德逊厘米,罗兹OE Swihart家乡。一年一度的阿勒格尼在非平衡metapopulation woodrats的生存。J哺乳动物。2016;97:1699 - 708。
亨德里克p基因流动和基因修复:佛罗里达豹作为案例研究。Conserv杂志。1995;9:996 - 1007。
Frankham r基因拯救小近交群体:荟萃分析显示大的和一致的基因流的好处。摩尔生态。2015;24:2610-8。
怀特利AR, Fitzpatrick SW、Funk WC Tallmon哒。遗传救援的救援。趋势生态另一个星球。2015;30:42-9。
Frankham r基因救助福利持续至少F3代,基于一个荟萃分析。Conserv杂志。2016;195:33-6。
Paetkau D, Strobeck c .微卫星分析黑熊种群的遗传变异。摩尔生态。1994;3:489 - 95。
Kalinowski美国多少等位基因/位点应该用于估计遗传距离吗?遗传。2002;88:62-5。
Tokarska M,马歇尔T, Kowalczyk R, Wojcik JM, Pertoldi C, Kristensen TN, et al .微卫星和SNP标记的有效性较低的血统和身份分析物种遗传多样性:欧洲野牛。遗传。2009;103:326-32。
Doyle JM Katzner TE,罗默GW,该隐JW,米尔萨普英航,麦金太尔CL,等。基因结构和可行性选择金鹰(Aquila chrysaetos泛北极分布),漫游的猛禽。Conserv麝猫。2016;17:1307-22。
代物第J,费尔南德斯N, Bruniche-Olsen Antonides J,柯南道尔J, San Miguel P, et al .灰鲸的特征(Eschrichtius南方)基因组和基因分型阵列基于候选基因的单核苷酸多态性。医学杂志。2017;232:186 - 97。
柯南道尔J,贝尔D,布鲁姆P,埃蒙斯G, Katzner T来普利L, et al .洞察系统发生学和人口结构的草原猎鹰(法尔科mexicanus)。BMC基因组学。2018;19:1-14。
豪泽L, Baird M, Hilborn R, Seeb LW, Seeb我。实证对比单核苷酸多态性微卫星的亲子关系和亲属关系的任务在一个野生红大麻哈鱼(雄鱼nerka人口)。摩尔生态Resour。2011; 11(增刊。1):150 - 61。
布坎南JW, Woronuk GN,侯爵FL,朗K,詹姆斯·圣Deobald H,等。分析验证和特定人群单核苷酸多态性在纯种血统面板和商业菜牛种群。J动画Sci。2017; 97:231-40。
Kaiser SA,泰勒SA,陈N,特TS、Bondra呃,韦伯斯特女士比较评估的SNP和微卫星标记分配出身在一个社会一夫一妻制的鸟。摩尔生态Resour。2017; 17:183 - 93。
张赵Thongda W, H, D, Jescovitch LN,刘M,郭X, SNP板等。发展作为一种新的工具来评估遗传多样性,人口结构和血统分析东部的牡蛎(Crassostrea virginica)。生物科技Mar》。2018; 20:385 - 95。
等待LP, Luikart G, Taberlet p .估计身份自然种群中基因型的概率:提醒和指导方针。摩尔生态。2001;10:249-56。
冯Thaden A、B Cocchiararo Jarausch,丛林h .评估SNP基因分型的非侵入性的收集野生动物样品使用微流控阵列。Sci众议员2017;7:10768。
C·冯·Thaden,诺瓦克,Tiesmeyer reiner TE,阿尔维斯PC,里昂洛杉矶,等。应用在野生动物基因组数据监控:开发指南的基因退化与单核苷酸多态性降低板样品。摩尔生态Resour。2020; 20:662 - 80。
坎贝尔M, Oakeson KF,扬德尔M, Halpert JR,迪林高产d基因组序列和注释的沙漠woodrat草案Neotoma 5种。基因组学数据。2016;9:58-9。
斯科菲尔德,杜尚J,拉金莱托,smyster TJ,道尔JM,尹浩然,斯科菲尔德。线粒体基因组的阿勒格尼Woodrat (Neotoma高地”)。线粒体DNA部分b . 2018; 3:256-8。
Ruegg KC,安德森EC,帕克斯顿KL Apkenas V,老挝,Siegel RB, et al .映射迁移在songbird使用高分辨率遗传标记。摩尔生态。2014;23:5726-39。
Bekkevold D, Helyar SJ, Limborg太,尼尔森EE, Hemmer-Hansen J,克劳森,et al .起源相关的基因标记可以分配在一个弱结构化鱼,大西洋鲱鱼。冰J Mar Sci . 2015; 72:1790 - 801。
Malenfant RM, Coltman DW,戴维斯CS。设计的9 k illumina公司BeadChip北极熊(北极熊从RAD)和转录组测序。摩尔生态Resour。2015; 15:587 - 600。
她Ferchaud l SH, Bekkevold D,剑J,妞妞Y,汉森毫米。低密度分析SNP数组微分选择在淡水和海洋的数量threespine棘鱼(Gasterosteus aculeatus)。BMC基因组学》2014。https://doi.org/10.1186/1471 - 2164 - 15 - 867。
Limborg太Helyar SJ,德Bruyn M,泰勒小姐,尼尔森EE,奥格登R,等。环境选择transcriptome-derived snp在高基因流动海洋鱼,大西洋鲱鱼(Clupea harengus)。摩尔生态。2012;21:2355-6。
代物第码,代物第农协。估计全基因组的杂合性使用分子标记。J在这里。2005;96:85-8。
Roffler GH,史密斯Amish SJ,年代,Cosart泰德,kardo m . SNP在候选人适应性基因发现使用外显子捕获在一个畅所欲言的高山有蹄类动物。摩尔生态Resour。2016; 16:1147 - 64。
艾尔米塔什霍夫曼霁,辛普森F,大卫·P JM, Kuiken T,索恩河畔,等。高通量测序揭示自然群体近交衰退。《美国国家科学院刊。2014;111:3775 - 80。
Crandall ED Clemento AJ,加尔萨JC,安德森EC,加尔萨JC,安德森EC。基因的单核苷酸多态性基线评估股票的识别大鳞大麻哈鱼(雄鱼tshawytscha在加利福尼亚海流大海洋生态系统。鱼牛。2014;112:112-30。
Edmands s之间进退两难:近亲繁殖和保护远系繁殖的相对风险评估和管理。摩尔生态。2007;16:463 - 75。
Frankham R, Ballou JD,埃尔德里奇MDB,花边RC,罗尔斯K,杜先生,等。远系繁殖的概率预测抑郁。Conserv杂志。2011;25:465 - 75。
Allison LJ斯科特•PA KJ, Averill-Murray RC,布拉德利SH。个人杂合性预测易位成功威胁沙漠陆龟。科学。2020;370:1086-9。
Hufford K,大杯美国植物生态型:遗传分化时代的生态修复。趋势生态另一个星球。2003;18:147-55。
Lenormand t基因流和自然选择的限制。趋势生态另一个星球。2002;17:183-9。
戴维斯MM, smyster TJ,约翰逊SA,杜尚J,拉金莱托,Swihart RK, et al .生殖成功captive-reared阿勒格尼Woodrats (Neotoma高地”)释放到基因萎缩的人口。Conserv麝猫。2021;22:903-12。
Lotsander, Hasselgren M, Larm M,瓦伦J Angerbjorn,诺尔k低持久性的跨代遗传救援北极狐(Vulpes lagopus)。J在这里。2021;112:276 - 85。
Gustafson KD,维氏TW,博伊斯WM,欧内斯特HB。一个单一的人口流动增强了天生的彪马的遗传多样性。R Soc Sci开放。2017;4:1-12。
维拉C, Sundqvist AK, FlagstadØ,Seddon J, Bjornerfeldt年代,Kojola我,et al。救援的严重瓶颈狼(犬属红斑狼疮)由一个移民人口。Proc R Soc b . 2003; 270:91-7。
亨德里克PW,彼得森RO Vucetich LM,亚当斯JR Vucetich农协。遗传在皇家岛狼救援:遗传分析和人口的崩溃。Conserv麝猫。2014;15:1111-21。
亨德里克P,罗宾逊J,彼得森R, Vucetich J .遗传学和灭绝和皇家岛狼的例子。动画Conserv。2019; 22:302-9。
罗宾逊是的,莱科宁J, Vucetich LM, Vucetich是的,彼得森RO, Lohmueller KE,等。基因签名的广泛的皇家岛狼,近亲繁殖人口灭绝的阈值。2019年Sci放置。https://doi.org/10.1126/sciadv.aau0757。
Stoffel马,卑微的E, Paijmans AJ,阿塞维多。怀特豪斯K, Chilvers提单,迪克森B, et al。人口历史和遗传多样性在鳍脚类动物受到人类开发、生态和生活史。Nat Commun。2018;六枝。
琼斯C,高贵的L,琼斯J, Tegelstrom H,组织G,贝瑞r .微分男性基因成功决定基因流动实验操纵鼠标的数量。Proc R Soc b . 1995; 260:251-6。
Ingvarsson PK,怀特洛克MC。杂种优势的有效迁移率增加。Proc R Soc b . 2000; 267:1321-6。
年代,Heinsohn R,艾伯特D, Legge Peakall R .遗传学证据相反色Eclectus鹦鹉合作一妻多夫制。动画Behav。2007; 74:1047-54。
Saccheri IJ, Brakefield点。迅速蔓延的移民基因组近交的人口。Proc R Soc b . 2002; 269:1073-8。
Allendorf弗兰克-威廉姆斯,Hohenlohe P Luikart g .基因组学和保护遗传学的未来。Nat牧师遗传学。2010;11:697 - 709。
Kanine JM, Kierepka EM Castleberry某人,Mengak MT, Nibbelink NP,格伦TC。景观异质性的影响功能连通性的阿勒格尼woodrats (Neotoma高地”维吉尼亚州的)。Conserv麝猫。2018;19:1259 - 68。
博尔格,Lohse M, Usadel b Trimmomatic:灵活的微调Illumina公司序列数据。生物信息学。2014;30:2114-20。
辛普森JT,黄K,杰克曼SD,史肯我,琼斯澳博,Birol i深渊:简称并行汇编程序读取序列数据。基因组研究》2009;19:1117-23。
西芒FA,沃特豪斯RM,埃尼迪斯P Kriventseva EV, Zdobnov EM。车身:评估基因组组装和注释与单副本直接同源完整性。生物信息学。2015;31:3210-2。
Nishimura O, Hara Y, Kuraku s gVolante标准化完整性评估基因组和转录组的组件。生物信息学。2017;33:3635-7。
Cantarel提单,Korf我罗伯SMC Parra G,罗斯E,摩尔B, et al。制造商:一个易于使用的新兴模式生物的基因组注释管道设计。基因组研究》2008;18:188 - 96。
柯南道尔JM Katzner TE, PH值,霁Y, Wijayawardena BK,代物第农协。广泛的基因组序列顶端食肉动物,金鹰(Aquila chrysaetos)。PLoS ONE。2014;9:20-2。
Korf即基因组的基因发现小说。BMC生物信息学。2004;5:59。
李H,杜宾r .快速和准确的短阅读符合burrows - wheeler变换。生物信息学。2009;25:1754-60。
van der Auwera GA, Carneiro莫,哈特尔C,府绸R, G del天使,Levy-Moonshine,等。从fastQ数据调用高信任度变体:基因组分析工具包管道的最佳实践。当前Protoc Bioinf。2013; 43:1-33。
DePristo马,银行E,府绸R,盖瑞K,马奎尔JR,哈特尔C, et al。变化的框架使用下一代DNA测序数据发现和基因分型。Nat麝猫。2011;43:491-8。
昆兰AR,大厅IM。BEDTools :一套灵活的工具来比较基因组的特性。生物信息学。2010;26:841-2。
王Cingolani P,普氏,L,浣熊,阮T,王L,等。一个程序注释和单核苷酸多态性的影响,预测SnpEff:基因的单核苷酸多态性黑腹果蝇应变w1118;iso-2 iso-3。飞。2012;6:1-13。
Thorvaldsdottir H,罗宾逊J, Mesirov J .整合基因组学查看器(进口):高性能基因组数据可视化和探索。短暂的Bioinform。2013; 14:178 - 92。
Caughley g .脊椎动物的数量分析。霍博肯:威利;1977年。
约翰逊SA。阿勒格尼woodrat重新评估(Neotoma高地”在印第安纳州。印第安纳州。2002;国家科学56 - 66。
Mengak M, Butchkoski C, D樵夫,约翰逊美国教训woodrat种群的长期监测。莱特:贝利J, J,编辑。阿勒格尼Woodrat:生态、减少物种的保护和管理。纽约:施普林格;2008年。109 - 31页。
克莱顿·d·snpStats: SnpMatrix和XSnpMatrix类和方法。2014年。
Kalinowski圣,锥形ML,马歇尔TC。修改计算机程序CERVUS如何适应基因分型错误增加亲子鉴定任务的成功。摩尔生态。2007;16:1099 - 106。
普里查德JK,史蒂芬斯M,唐纳利p .茎人口结构使用基因型数据的推理。遗传学。2000;155:945-59。
唐尼尔D,史蒂芬斯M, Pritchard JK。茎推理的人口结构使用基因型数据:连接位点等位基因频率和相关。遗传学。2003;164:1567 - 87。
Castleberry某人,国王TL,木PB,福特WM。微卫星DNA分析人口结构的阿勒格尼woodrats (Neotoma高地”)。J哺乳动物。2002;83:1058 - 70。
smyster TJ,约翰逊SA、页面路、哈德逊厘米,罗兹OE。使用实验易位的阿勒格尼woodrat破译因果代理的下降。Conserv杂志。2013;27:752 - 62。
伯爵D, VonHoldt BM。收割机结构:一个网站和程序可视化结构输出和实现Evanno方法。Conserv麝猫Resour。2011; 4:359 - 61。
杰可布森M, Kopelman NM, Mayzel J,罗森博格NA, Mayrose i CLUMPAK:一个程序识别集群模式和包装在k·摩尔生态Resour人口结构推断。2015;15:1179 - 91。
Peakall R, Smouse PE。GenAlEx 6, 5:遗传分析在excel中人口遗传软件教学和研究更新。生物信息学。2012;28:2537-9。
代物第J, Avise JC。微卫星海洋的变化,淡水和溯河产卵的鱼类与其他动物相比。J鱼杂志。2000;56:461 - 73。
Eo年代,柯南道尔J,代物第J .遗传多样性在鸟类与体重和栖息地类型相关联。黑旋风。2011;283:220-6。
柯南道尔JM,黑客CC,小威洛比,《M,代物第农协。哺乳动物遗传多样性的栖息地,体型,营养类,和保护状态。J哺乳动物。2015;96:564 - 72。
小威洛比,《M, Wijayawardena BK,金布尔SJA,霁Y,费尔南德斯NB,等。基因多样性的减少威胁脊椎动物和新建议关于保护世界自然保护联盟排名。Conserv杂志。2015;191:495 - 503。
小威洛比,《M, Wijayawardena BK、羊肉MC,金布尔SJA,霁Y, et al。生物群系和迁徙行为显著影响脊椎动物遗传多样性。J林Soc杂志。2017;121:446-57。
Schunter C,加尔萨JC,麦克弗森E,帕斯卡·m·SNP发展RNA-seq数据nonmodel鱼:有多少人需要准确的等位基因频率的预测?摩尔生态Resour。2014; 14:157 - 65。
里德Baetscher DS, Hasselman DJ, K, Palkovacs EP,加尔萨JC。发现和描述的单核苷酸多态性在两个溯河产卵的alosine鱼类的保护问题。生态另一个星球。2017;7:6638-48。
里德K Palkovacs EP Hasselman DJ, Baetscher D, Kibele J, Gahagan B,等。基因种群结构的综合评价与单核苷酸多态性数据河溯河产卵的鲱鱼。鱼杂志2018;206:247-58。
戴维斯CD,加尔萨JC,银行。识别多种遗传学上截然不同的奇努克鲑鱼的数量(雄鱼tshawytscha)在一个小沿海分水岭。环境生物鱼类。2017;100:923-33。
福特MJ,默多克AR,休斯女士,Seamons TR,拉胡德s养殖历史强烈影响自然产卵的成功孵化器虹鳟(雄鱼mykiss)。PLoS ONE。2016;11:1-20。
Katzner TE,纳尔逊DM, Braham妈,柯南道尔JM,费尔南德斯NB, Duerr AE, et al。金鹰死亡和当地风能发电的大陆范围内的后果。Conserv杂志。2017;31:406-15。
Mathur年代,Tomeček JM, Heniff Luna R,代物第农协。遗传侵蚀的证据在北美游戏周边人口鸟:Montezuma鹌鹑(Cyrtonyx montezumae)。Conserv麝猫。2019;20:1369 - 81。
特里B,莉丝K,沈J,何鸿燊J,张载L, 1 g的多个司机年际牡蛎结算和招聘低切萨皮克湾。Conserv麝猫。2019;20:1057 - 71。
特里BD。牡蛎礁连接通过人口遗传分析推断。2015年。
天《GQ》, Ng J, Oldt射频,霍顿PW,史密斯DG Kanthaswamy s dna测定祖先在猕猴猕猴(猕猴属fascicularis)。J是Assoc实验室动物科学。2018;57:432-42。
确认
这项工作用了极端的科学和工程发现环境(XSEDE),由美国国家科学基金会支持号码aci - 1548562。具体地说,这项工作用彗星通过分配TG-BIO170095圣地亚哥超级计算机中心。作者感谢杰弗里·拉金准备资金支持这项工作的建议;阿曼达Menasion, Sharon Petzinger戴夫•肯尼Kathleen LoGuidice爱兰歌娜米切尔,凯特·豪斯曼帮忙收集n高地”样品和跟踪遥测和摄像机的转移个人;法灵顿和凯莉•布兰德,奇迹戴维斯,希瑟,曼迪斯科菲尔德和伊莱Seeman与SNP面板发展援助,DNA提取和量化和微卫星基因分型。
资金
这项研究受到了印第安纳州的自然资源,新泽西DEP鱼类和野生动物的濒危物种和Nongame项目,物保护鱼类和野生动物服务野生动物恢复计划,宾夕法尼亚州游戏委员会,美国大自然保护协会,普渡大学,陶森大学费舍尔科学和数学学院。
作者信息
作者和联系
贡献
JD、广发、GGT、套和托拉斯的构思和设计研究。JD, GF,厘米,BW和GGT收集现场数据和样本。MMG、SK、套和托拉斯进行提取DNA, DNA量化和SNP基因分型。套和托拉斯进行全基因组测序,组装基因组SNP面板和设计草案。MMG GF, SK和托拉斯写作努力和所有作者(MMG GF, JD, SK,厘米,套,GGT, BW和托拉斯)审查、修订,评论和批准的最终版本的手稿。
相应的作者
道德声明
伦理批准和同意参与
所有方法和协议是一个机构和/或许可委员会批准,按照指导方针和有关规定进行。标准住捕获和样本收集协议被用于所有情况和已批准的机构动物保健和使用委员会(IACUC)宾夕法尼亚印第安纳大学,印第安纳州,PA(协议# 10 - 1617和10 - 1920)和普渡大学西拉法叶,(协议# 1111000236和1111000236)。所有捕获和印第安纳州组织样本收集,完成了新泽西州和宾夕法尼亚州宾州游戏委员会,新泽西的鱼类和野生动物的濒危物种和Nongame项目和印第安纳州自然资源部门(机构由州法律规定审查和执行所有监控和允许的话)。所有被困在俄亥俄州和组织样本收集完成了WildWork俄亥俄州的许可部门的野生动物(科学收集允许# 21 - 181)。这项工作要经国家环境政策法案,这是由美国鱼类和野生动物服务。本研究报告按照到达的指导方针。
同意出版
不适用。
相互竞争的利益
作者声明他们没有利益冲突。
额外的信息
出版商的注意
施普林格自然保持中立在发表关于司法主权地图和所属机构。
补充信息
额外的文件1:
微卫星基因型woodrats采样2009年,2011年和2012年在栅栏,新泽西。电子表格标记为“原始数据”,“数据GenAlEx”包括28个人的基因型在11个微卫星位点基因分型。这些人有相对较低的遗传变异,表明等位基因的数量(法新社和榴弹炮电子表格),观察到的杂合性和预期的杂合性(电子表格六)。
额外的文件2:
摘要统计信息与核基因组测序,组装和注释。
额外的文件3:
SNP基因型woodrats采样在印第安纳州、新泽西州、俄亥俄州和宾夕法尼亚州。电子表格标记为“原始数据”包括318人的基因型在134个SNP位点基因分型。
额外的文件4:
身份(PID)的概率之间的关系,兄弟姐妹之间身份的概率(PIDsib)和SNP或微卫星位点的基因的数量。
额外的文件5:
比较微卫星和SNP-based遗传变异。
权利和权限
开放获取本文是基于知识共享署名4.0国际许可,允许使用、共享、适应、分布和繁殖在任何媒介或格式,只要你给予适当的信贷原始作者(年代)和来源,提供一个链接到创作共用许可证,并指出如果变化。本文中的图片或其他第三方材料都包含在本文的创作共用许可证,除非另有说明在一个信用额度的材料。如果材料不包括在本文的创作共用许可证和用途是不允许按法定规定或超过允许的使用,您将需要获得直接从版权所有者的许可。查看本许可证的副本,访问http://creativecommons.org/licenses/by/4.0/。知识共享公共领域奉献豁免(http://creativecommons.org/publicdomain/zero/1.0/)适用于数据可用在这篇文章中,除非另有说明在信贷额度的数据。
关于这篇文章
引用这篇文章
Muller-Girard, M。家禽,G。,Duchamp, J.et al。小说SNP分析揭示遗传变异性增加和丰富易位后残余阿勒格尼woodrat人口。BMC生态Evo22137 (2022)。https://doi.org/10.1186/s12862 - 022 - 02083 - w
收到了:
接受:
发表:
DOI:https://doi.org/10.1186/s12862 - 022 - 02083 - w
关键字
- 生殖斜
- 近亲繁殖
- 基因修复
- 近交衰退
- 易位
- 景观遗传学
- 传播